Was sind Bioassays und was macht LuminoGen besonders?
Quellen sind im Fließtext durch eckige Klammern [ ] gekennzeichnet und am Ende der Seite aufgeführt
LuminoGen in sechs Sätzen erklärt
1) Bioassays sind lebende Zellen, die über Reportergene messbare Signale erzeugen
2) Bioassays detektieren biologisch aktive Stoffmengen
3) Unter den gängigen Reportergenen werden Luciferasen immer beliebter
4) Luciferasen benötigen den „Brennstoff“ Luciferin, um Licht zu erzeugen
5) Das Problem: Die vielen Nachteile der Abhängigkeit von externem Luciferin
6) Die Lösung: LuminoGen-Hefen synthetisieren ihr eigenes Luciferin
1) Bioassays sind lebende Zellen, die über Reportergene messbare Signale erzeugen
Zellen wie Bakterien, Pilze oder humane und tierische Zellen als auch mehrzellige Lebewesen wie z.B. Pflanzen besitzen von sich aus verschiedene sensorische Systeme auf molekularem Level, die es den Zellen ermöglichen, auf veränderte Umweltbedingungen oder bestimmte Reize zu reagieren (Abbildung 1).
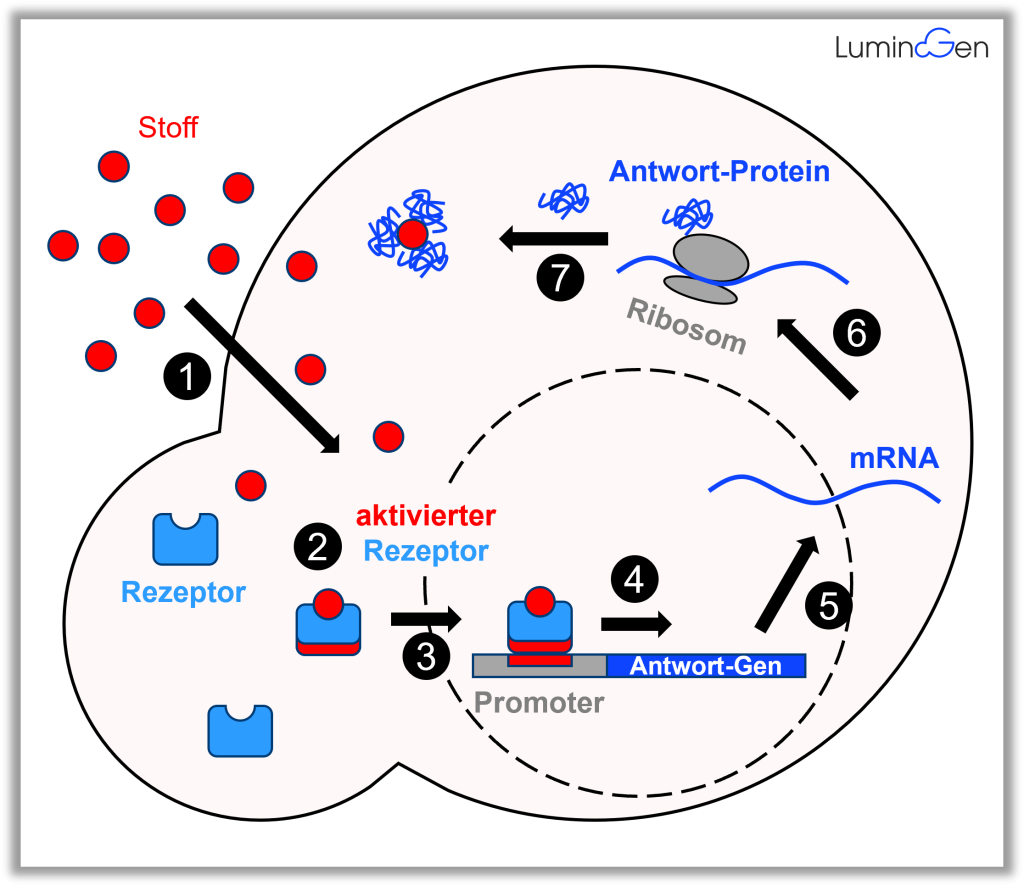
Diese natürlichen molekularen Signalwege kann man mit Hilfe von Reportergenen abhören und damit sichtbar und messbar machen. Reportergene werden dazu genetisch an einen molekularen Signalweg gekoppelt, sodass diese immer dann aktiviert werden, wenn auch der Signalweg in der Zelle aktiv wird.
Die Kopplung der Reportergene in den Signal- oder Stoffwechselweg einer Zelle erfolgt durch eine genetische Veränderung. In den meisten Fällen steht das Reportergen unter denselben Gen-aktivierenden Sequenzen (Promotoren) wie jene Gene in der Zelle, deren Aktivität gemessen werden soll (Abbildung 2).
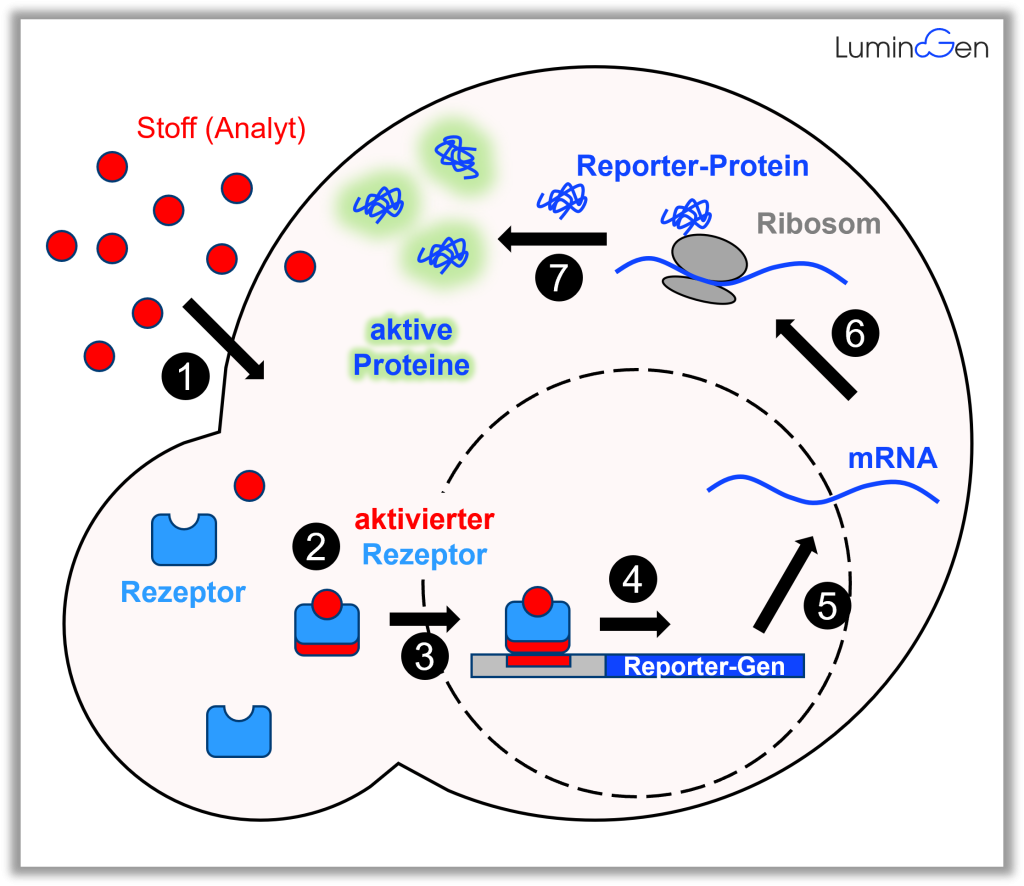
Durch das aktivierte Reportergen werden dann Reporter-Proteine in einer Zelle gebildet, deren Anwesenheit in der Zelle leicht von außerhalb messbar sind, z.B. weil sie durch UV-Licht anfangen zu fluoreszieren, oder weil diese Proteine zu einer Verfärbung führen, oder sogar Licht erzeugen.
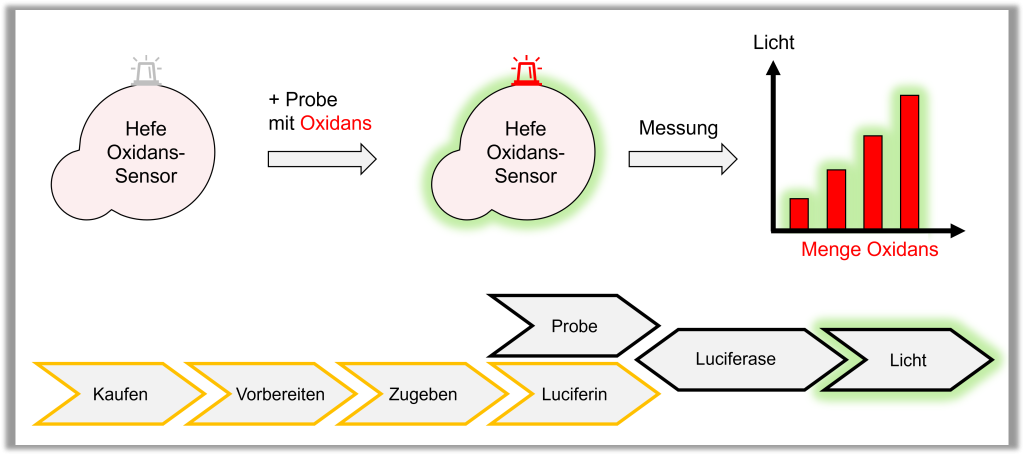
Ein einfaches Beispiel (Abbildung 3): Erfährt eine Zelle z.B. vermehrt oxidativen Stress durch äußere oder innere Einflüsse, dann werden in der Zelle Gene aktiviert, um diesem Stress entgegen zu wirken. Ein an ein solches Gen gekoppeltes Reportergen wird dann gleichzeitig aktiviert, wodurch ein Biosensor oder ein sogenannter Bioassay für oxidativen Stress gegeben ist. D.h. die Menge an oxidativem Stress, den eine Zelle erfährt, ist durch die Aktivität des Reportergens messbar, also z.B. durch die Menge an Farbe oder Licht, die die Zelle erzeugt.
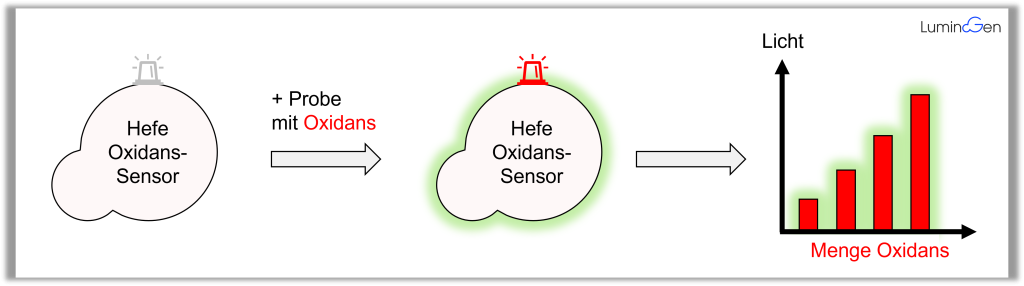
2) Bioassays detektieren biologisch aktive Stoffmengen
Bioassays werden z.B. zur Detektion von hormonell aktiven Substanzen angewendet, die aus der Umwelt in Wasser, Lebensmittel oder Kosmetika gelangen können. Der Vorteil von Bioassays ist, dass biologisch wirksame Stoffe gemessen werden.
Durch physikalische oder chemische Proben lassen sich zwar Konzentrationen von Stoffen ermitteln, zu denen es Erfahrungswerte für giftige Konzentrationen gibt, aber ob eine Probe oder ein Stoffgemisch in Summe oder synergistisch einen biologischen Effekt hat, können physikalisch/chemische Methoden nicht erfassen.
3) Unter den gängigen Reportergenen werden Luciferasen immer beliebter
Reportergene werden durch Gentechnik in andere Organismen übertragen, um bestimmte zelluläre Aktivitäten zu messen. Eines der ersten Reportergene stammt aus dem sogenannten lac-Operon von dem Darmbakterium Escherichia coli. Das lac-Operon ist grob gesagt eine Gruppe von Genen, die zur Verwertung von Lactose (Milchzucker) aktiviert wird [1].
Eines dieser Gene namens lacZ codiert ein Protein namens β-Galactosidase. Dieses Protein ist ein Enzym, dass Zucker-Bindungen (glykosidische Bindungen) spaltet und normalerweise zur Spaltung des Zuckers Lactose dient. Wissenschaftler können dieses Gen als Reportergen nutzen, indem sie die Zellen, die dieses Gen tragen, vorher mit Farbstoffen mit glykosidischer Bindung füttern. Wird das Glykosid vom Farbstoff gespalten, ändert sich das Spektrum zu einer anderen Farbe, wobei Farbton und Farbmenge leicht messbar sind [1].
Ein ebenfalls bekanntes Reportergen ist das sogenannte Green fluorescent Protein (GFP), welches aus einer Qualle (Aequorea victoria) stammt. Das GFP-Protein fluoresziert grün, wenn es mit UV-Licht beleuchtet wird, und so auch die Zellen, die dieses Protein bilden [2].
In den letzten Jahren haben Luciferase-Gene als Reportergene immer höhere Beliebtheit erlangt (Abbildung 4). Luciferase-Proteine erzeugen Licht, wodurch Zellen mit diesen Genen leuchten. Das bekannteste Luciferase-Gen ist wahrscheinlich jenes aus Glühwürmchen, welches diesen Insekten ihr grünes Leuchten in der Nacht verleiht [3,4].
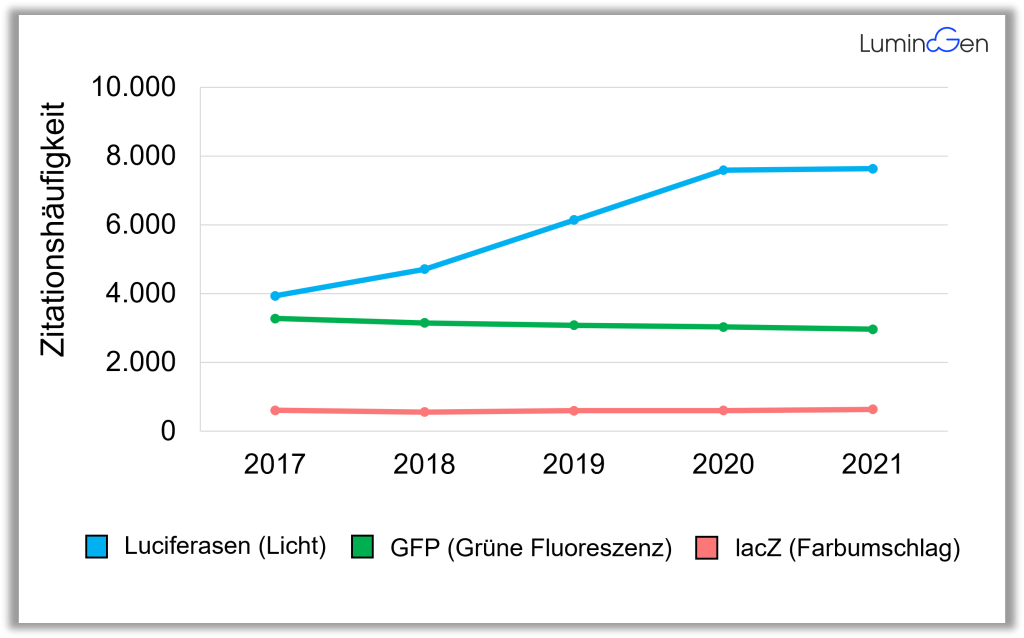
Luciferase-Gene sind allerdings auch bei Bakterien [5], Pilzen [6] , Würmern [7] und Seefedern [8] bekannt. Die Beliebtheit für Luciferase-Reportergene beruht darauf, dass das von den Luciferase-Proteinen produzierte Licht sehr sensitiv durch Photozellen messbar ist, wodurch sehr geringe Gen-Aktivitäten leicht nachweisbar sind. Das Prinzip ist ähnlich wie bei Solarzellen: Licht-Photonen treffen auf eine Oberfläche und erzeugen Strom. Dadurch lassen sich also sehr sensitive Bioassays konstruieren.
4) Luciferasen benötigen den „Brennstoff“ Luciferin, um Licht zu erzeugen
Ähnlich wie bei der β-Galactosidase brauchen Luciferasen ein sogenanntes Substrat, also einen Stoff, den sie als Enzym in ein Produkt umsetzen. Bei Luciferasen ist das Substrat das sogenannte Luciferin, welches man in diesem Kontext auch als notwendigen Brennstoff für die Bildung von Licht bezeichnen kann.
Die Glühwürmchen-Luciferase ist eine der am meisten verwendete, und früher wurde das Substrat für diese Luciferase (D-Luciferin) direkt aus Leuchtkäfern gewonnen. Mittlerweile wird es chemisch hergestellt [9,10] und zum Kauf angeboten. In der biotechnologischen Anwendung befinden sich derzeit drei Luciferin/Luciferase Systeme: aus der Seefeder (Renilla reniformis), aus Glühwürmchen (Photinus pyralis) und aus Bakterien (z.B. aus Photorhabdus luminescens).
Jedes System verwendet seine eigene Luciferase, die nur das jeweils dafür passende Luciferin zu Licht umsetzen kann. So kann z.B. die Glühwürmchen-Luciferase kein bakterielles Luciferin nutzen.
Das am häufigsten verwendete Luciferase-System ist das des Glühwürmchens, da es auch in Eukaryoten anwendbar ist und über die Zeit für die Analytik optimiert wurde. Beispielsweise wurden verschiedene Substrate und Luciferase-Varianten entwickelt, um die Lichtfarbe von grün in z.B. rot zu ändern und damit simultane Assays zu ermöglichen [11,12]. Ebenfalls ist die Interaktion von zwei Proteinen mit der sogenannten Split-Luciferase-Technik [13] messbar.
5) Das Problem: Die vielen Nachteile der Abhängigkeit von externem Luciferin
Wir haben an der Universität viele Assays pipettiert, um die unterschiedlichsten Substanzen mit Glühwürmchen-Reporter-Hefen zu untersuchen. Was uns an dem Glühwürmchen-Luciferase Assay gestört hat waren sämtliche mit D-Luciferin verbundenen Nachteile (Abbildung 5): ständige Preisvergleiche, um günstiges Luciferin zu erhalten, die Vorbereitung des Luciferins mit pH-eingestellten Lösungen, und letztlich die Zugabe zu jeder einzelnen Messung.
Damit in Verbindung verloren wir auch Zeit in der Assay-Planung durch die notwendige Kalkulation von Luciferin: wie viel Messvolumen muss ich für Luciferin einkalkulieren, und wie verdünne ich es am besten?
Um die kostbare Chemikalie möglichst effizient einzusetzen, haben wir auch öfters bereits angebrochene Lösungen wiederverwendet, wobei es immer die Unsicherheit gab, ob durch die Lagerung (Zeit und ggf. Einfrier- und Auftauzyklen) die anfängliche Qualität des Luciferins noch gegeben war.
Um dieses Problem zu lösen haben wir nach viel Literaturrecherche und Planung versucht, die bakterielle Biolumineszenz in Hefe zu übertragen. Übertragen wurde hierbei also nicht nur die bakterielle Luciferase, sondern auch sämtliche Gene für die bakterielle Luciferin-Synthese.
Da die Gene für die Synthese des Glühwürmchen-Luciferins bis dato (Stand: April 2023) noch nicht identifiziert wurden, waren die bereits publizierten Bakterien-Gene neben unserer eigentlichen Forschungsarbeit (nämlich der eigentlichen Durchführung von Bioassays) unsere einzige machbare Lösung, die keine zusätzliche Doktorarbeit erfordern würde. Unser damaliger Master-Student Michael Ries hat die Hefe dann aus der Planungsphase in einen leuchtenden Hefe-Prototypen verwirklicht.
6) Die Lösung: LuminoGen-Hefen synthetisieren ihr eigenes Luciferin und sparen dadurch Kosten- und Zeit
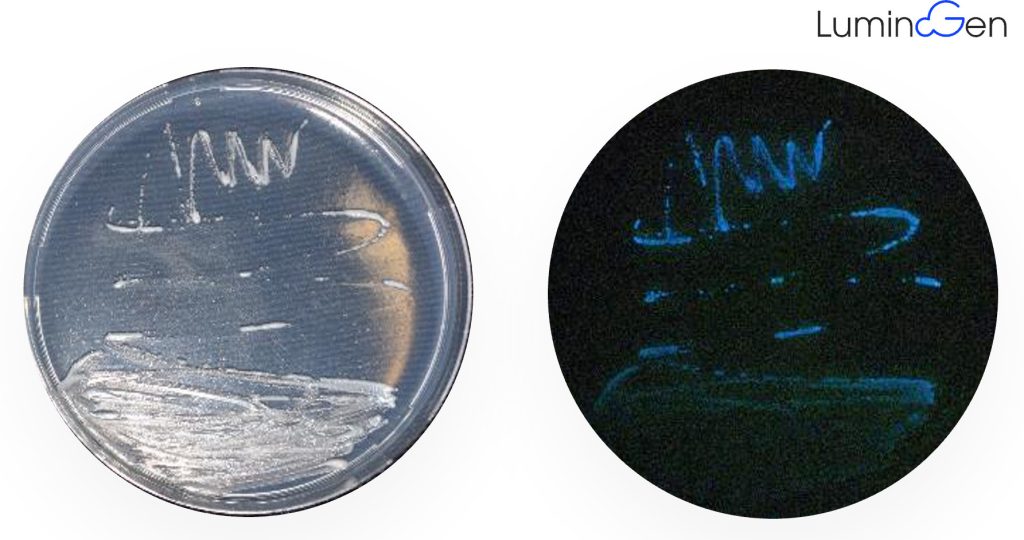
Wir haben den von Michael erstellten Hefeprototyp verschiedenen Tests unterzogen. Natürlich ist es bei Biolumineszenz immer eine spannende Frage, wie stark diese ausgeprägt wird, und ob und wie gut man diese sehen kann. Die blaue Biolumineszenz des LuminoGen-Hefeprototyps ist mit bloßem Auge bei Dunkelheit sehr gut sichtbar (Abbildung 6), wobei uns die Leuchtstärke sehr überrascht hat, weshalb wir wissen wollten, wie sich der Prototyp in einem direkten Vergleich mit einer Hefe mit Glühwürmchen-Luciferase schlägt.
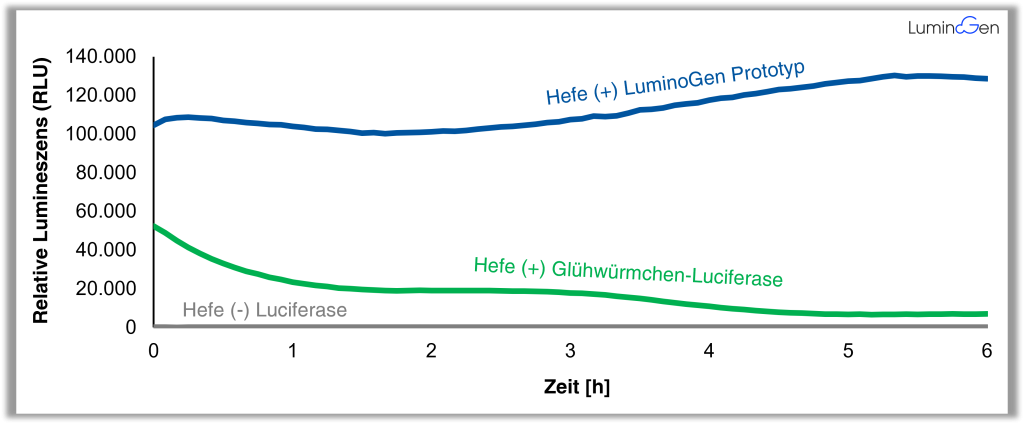
Für den Vergleich der neuen Technik mit dem Glühwürmchen-Luciferase-Reporter in Hefe haben wir beide Luciferase-Systeme unter dieselben genetischen Regulatoren gestellt, damit die Genaktivität beider Systeme vergleichbar ist. Ebenso haben wir dasselbe genetische Trägermaterial (Plasmide) verwendet, um die Gene in Hefe zu überführen. Dadurch ist auch die Kopienzahl der genetisch übertragenen Information gleich hoch.
Unsere Experimente zeigten eindeutig und reproduzierbar, dass unser erfundener Prototyp unter diesen Vergleichsbedingungen etwa 2,5-fach mehr Licht als die Glühwürmchen-Luciferase erzeugt. Zusätzlich weist unser Prototyp eine höhere Ausdauer auf, was zu erwarten war, da er den notwendigen Brennstoff Luciferin selbst synthetisiert, wohingegen das Substrat im Glühwürmchen-System limitiert ist (Abbildung 7). Der Anstieg der Biolumineszenz nach vielen Stunden ist durch das Wachstum zu erklären: mehr Zellen erzeugen auch mehr Licht. Gleichermaßen verbrauchen mehr Zellen auch mehr Luciferin, wie an der Messkurve für die Glühwürmchen-Luciferase zu beobachten ist.
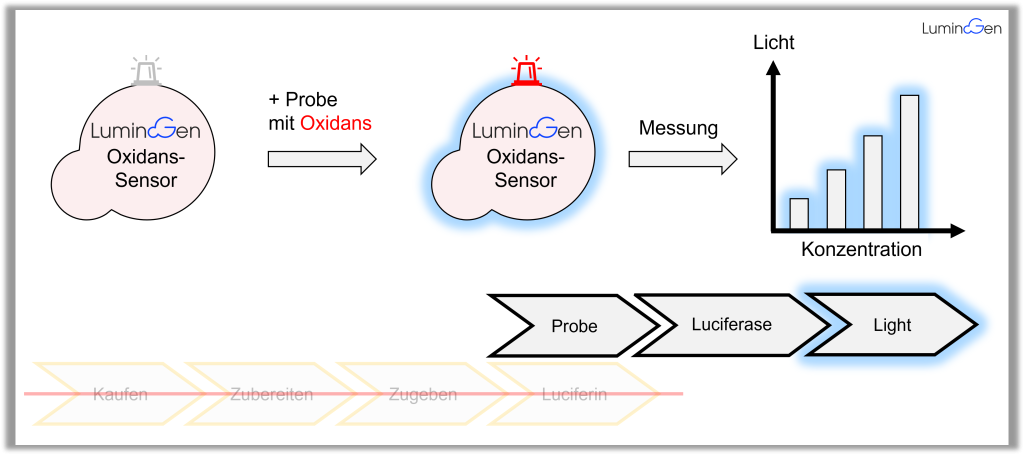
Mit diesen Messergebnissen und der Unabhängigkeit von Luciferin werden dadurch Bioassays in dem notwendigen Zeit- und Kostenaufwand erheblich vereinfacht (Abbildung 8).
Bioassays werden in Hochdurchsatzanalysen verwendet, was bedeutet, dass viele Proben in einem Durchlauf gemessen werden. Für gewöhnlich findet dies im Mikrotiter-Plattenmaßstab statt, wobei eine Platte mit 96 Proben bestückt werden kann. Eine solche Platte wird dann in einen Platereader eingesetzt, der mit einem Messkopf über jede Vertiefung fährt, und die Signale der darin befindlichen Zellen misst (Abbildung 9).

In diesem Maßstab können an einem Tag also mehrere hundert oder mehrere tausend Messungen durchgeführt werden. Solche Hochdurchsatzverfahren (engl. High Throughput Screening, HTS) werden z.B. im Umwelt-Monitoring angewandt, wobei regelmäßig Proben auf chemische Kontaminationen geprüft werden, oder auch im Produkt-Monitoring oder bei der Entwicklung pharmakologischer Wirkstoffe.
Quellenangabe
- Shuman, H. A.; Silhavy, T. J. The Art and Design of Genetic Screens: Escherichia coli. Nature Reviews Genetics 2003, 4, 419.
- Tsien, R. Y. The Green Fluorescent Protein. Annu. Rev. Biochem. 1998, 67 (1), 509–544. https://doi.org/10.1146/annurev.biochem.67.1.509.
- de Wet, J. R.; Wood, K. V.; Helinski, D. R.; DeLuca, M. Cloning of Firefly Luciferase cDNA and the Expression of Active Luciferase in Escherichia coli. Proc Natl Acad Sci USA 1985, 82 (23), 7870. https://doi.org/10.1073/pnas.82.23.7870.
- de Wet, J. R.; Wood, K. V.; DeLuca, M.; Helinski, D. R.; Subramani, S. Firefly Luciferase Gene: Structure and Expression in Mammalian Cells. Mol. Cell. Biol. 1987, 7 (2), 725. https://doi.org/10.1128/MCB.7.2.725.
- Engebrecht, J.; Nealson, K.; Silverman, M. Bacterial Bioluminescence: Isolation and Genetic Analysis of Functions from Vibrio fischeri. Cell 1983, 32 (3), 773–781.
- Ke, H.-M.; Tsai, I. J. Understanding and Using Fungal Bioluminescence – Recent Progress and Future Perspectives. Current Opinion in Green and Sustainable Chemistry 2022, 33, 100570. https://doi.org/10.1016/j.cogsc.2021.100570.
- Rodionova, N. S.; Rota, E.; Tsarkova, A. S.; Petushkov, V. N. Progress in the Study of Bioluminescent Earthworms. Photochemistry and Photobiology 2017, 93 (2), 416–428. https://doi.org/10.1111/php.12709.
- Lorenz, W. W.; McCann, R. O.; Longiaru, M.; Cormier, M. J. Isolation and Expression of a cDNA Encoding Renilla reniformis Luciferase. Proceedings of the National Academy of Sciences 1991, 88 (10), 4438–4442. https://doi.org/10.1073/pnas.88.10.4438.
- White, E. H.; McCapra, Frank.; Field, G. F. The Structure and Synthesis of Firefly Luciferin. J. Am. Chem. Soc. 1963, 85 (3), 337–343. https://doi.org/10.1021/ja00886a019.
- Oba, Y.; Yoshida, N.; Kanie, S.; Ojika, M.; Inouye, S. Biosynthesis of Firefly Luciferin in Adult Lantern: Decarboxylation of ʟ-Cysteine Is a Key Step for Benzothiazole Ring Formation in Firefly Luciferin Synthesis. PLOS ONE 2014, 8 (12), e84023. https://doi.org/10.1371/journal.pone.0084023.
- Mamaev, S. V.; Laikhter, A. L.; Arslan, T.; Hecht, S. M. Firefly Luciferase: Alteration of the Color of Emitted Light Resulting from Substitutions at Position 286. J. Am. Chem. Soc. 1996, 118 (30), 7243–7244. https://doi.org/10.1021/ja961053c.
- Jathoul, A. P.; Branchini, B. R.; Anderson, J. C.; Murray, J. A. H. A Higher Spectral Range of Beetle Bioluminescence with Infraluciferin. Frontiers in Bioengineering and Biotechnology 2022, 10.
- Ozawa, T.; Kaihara, A.; Sato, M.; Tachihara, K.; Umezawa, Y. Split Luciferase as an Optical Probe for Detecting Protein−Protein Interactions in Mammalian Cells Based on Protein Splicing. Anal. Chem. 2001, 73 (11), 2516–2521. https://doi.org/10.1021/ac0013296.
- Bancos, I.; Bida, J. P.; Tian, D.; Bundrick, M.; John, K.; Holte, M. N.; Her, Y. F.; Evans, D.; Saenz, D. T.; Poeschla, E. M.; Hook, D.; Georg, G.; Maher, L. J., III. High-Throughput Screening for Growth Inhibitors Using a Yeast Model of Familial Paraganglioma. PLOS ONE 2013, 8 (2), e56827. https://doi.org/10.1371/journal.pone.0056827.
Förderer und Partner
Laufende und vergangene Förderung
- Das Projekt LuminoGen wurde im Rahmen des EXIST-Programms durch das Bundesministerium für Wirtschaft und Klimaschutz und den Europäischen Sozialfonds gefördert. (April 2023 – April 2024)
- BIO.NRW Start Up Paket für die EFIB-Tagung in Rotterdam (November 2023), hier geht es zu dem zugehörigen Beitrag von BIO.NRW
